1.3. Estructuras de las proteínas
Pese a la rigidez de los enlaces peptídicos, una proteína podría, en principio, adoptar múltiples conformaciones —disposiciones espaciales de sus átomos que pueden lograrse sin romper ningún enlace covalente—, ya que los enlaces con el carbono α son simples y, por tanto, girarían libremente en respuesta al movimiento térmico ambiental. Sorprendentemente, en las condiciones biológicas normales de pH y de temperatura, las cadenas polipeptídicas suelen poseer una única conformación nativa, que además es la responsable de su función biológica: una alteración de dicha conformación nativa significaría una pérdida de funcionalidad.
Entender esta aparente paradoja pasa por estudiar con detalle la estructura de las proteínas que, por comodidad, dividiremos en varios niveles de complejidad creciente:
- Estructura primaria
Se refiere a la secuencia lineal de aminoácidos unidos por enlaces peptídicos. Las cadenas peptídicas están polarizadas, esto es, tienen dos extremos bien definidos: un extremo N-terminal, que presenta el grupo amino libre (por convenio suele escribirse a la izquierda), y un extremo C-terminal, que tiene el grupo carboxilo libre (a la derecha). Un ejemplo de secuencia peptídica sería:
H2N–Asp–Phe–Met–Cys–Pro–Lys–Asn–Ala–His–COOH La estructura primaria es de gran importancia porque de ella dependen en buena medida todos los demás niveles estructurales. La alteración de la secuencia de aminoácidos de un polipéptido dará lugar a una proteína distinta que podría realizar una función diferente o —mucho más frecuentemente— perder su actividad biológica. Incluso dos polipéptidos serán diferentes, aún teniendo los mismos aminoácidos, si éstos están situados en un orden distinto.
- Estructura secundaria
Este término alude a la conformación local de algunas regiones del polipéptido; es decir, a la disposición regular de los aminoácidos en un segmento de la cadena polipeptídica, en la que cada aminoácido se relaciona espacialmente con sus vecinos de la misma manera. Una sola proteína podrá exhibir distintas estructuras secundarias en diferentes tramos de su cadena polipeptídica; dependerá sobre todo de las cadenas laterales R, ya que su tamaño y su carga pueden imponer restricciones al libre giro en torno a los enlaces no peptídicos. Por tanto, solo serán estables aquellas estructuras secundarias en las que los átomos de la proteína puedan acomodarse en el espacio delimitado por las citadas restricciones, de modo tal que se formen enlaces de hidrogeno intracatenarios entre los grupos –C=O y –NH de diferentes enlaces peptídicos. En ausencia de tales interacciones estabilizadoras habrá un enrollamiento aleatorio o cadena estadística, sin estructuras secundarias bien definidas.
Solo unas cuantas estructuras secundarias están distribuidas ampliamente. Entre ellas destacan la hélice alfa, la hélice de colágeno, la lámina beta plegada y los giros beta.
-
Hélice alfa o α-hélice. La cadena de aminoácidos se enrolla sobre sí misma, en forma de hélice que gira en sentido dextrógiro (hacia la derecha), debido a la especial disposición en que se van orientando los aminoácidos al enlazarse, y que determina que cada plano que contiene un enlace peptídico realice un giro de unos 120º respecto al plano anterior. Los enlaces de hidrógeno que se establecen entre el –C=O de un aminoácido y el –NH del cuarto aminoácido siguiente son los responsables de estabilizar la estructura. La α-queratina, muy abundante en las células epidérmicas, es un ejemplo de proteína que solo tiene estructura de α-hélice.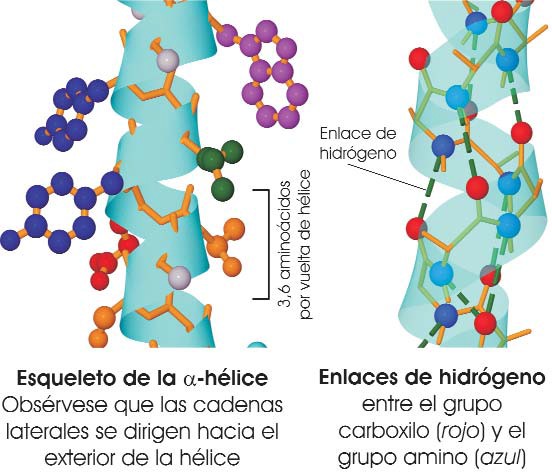
Esquema de la α-hélice de una proteína de un inovirus (fuente ASH). - Hélice del colágeno. A diferencia de la α-hélice, se trata de una hélice levógira —que gira a la izquierda—. Además, la hélice está más distendida, debido fundamentalmente a la abundancia de los aminoácidos prolina e hidroxiprolina, con cadenas laterales muy voluminosas, que dificultan la formación de enlaces de hidrógeno intracatenarios.
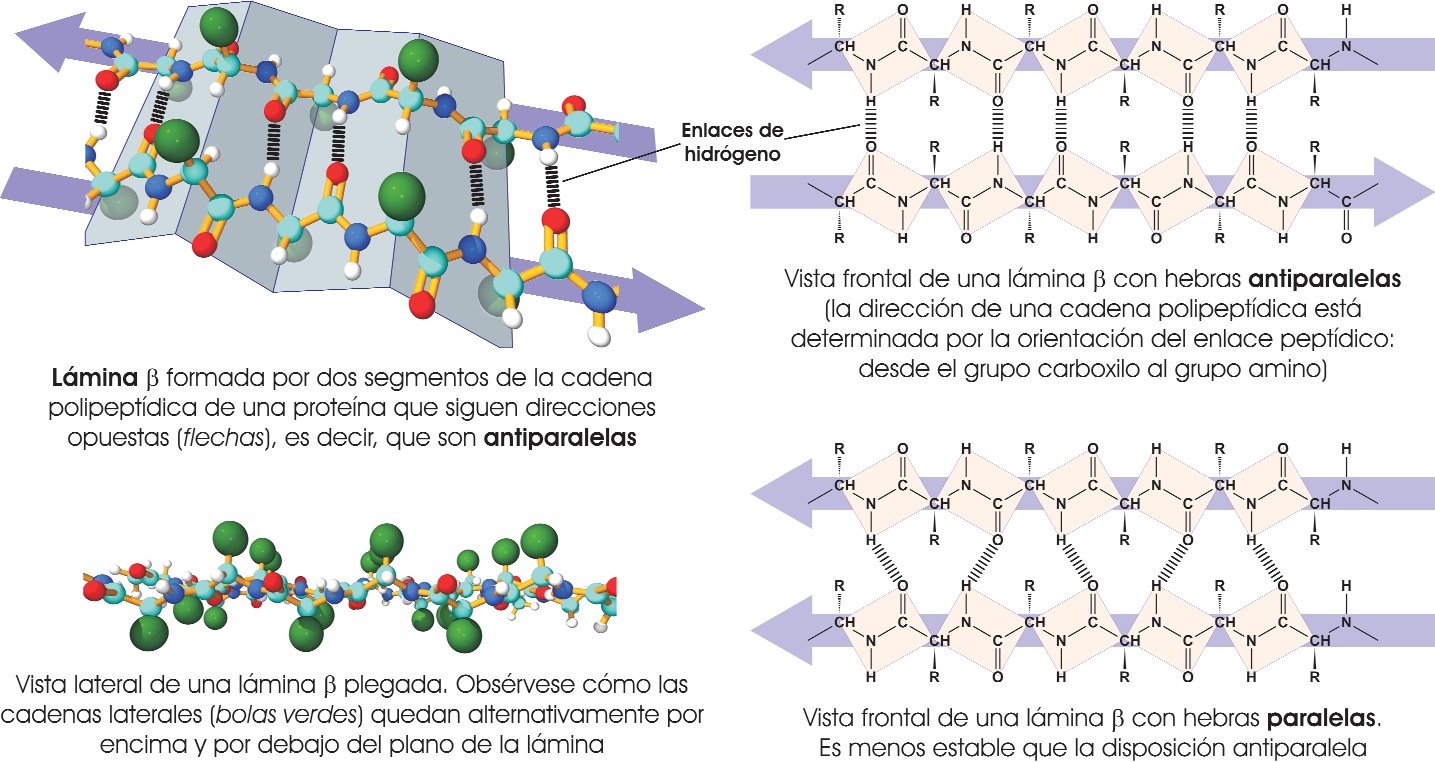
- Lámina β. Es una conformación más relajada en forma de hoja plegada en zigzag, en la que varios segmentos extendidos, de 5 a 8 aminoácidos de longitud, se disponen de manera adyacente y se unen mediante enlaces de hidrógeno [véase la ilustración a continuación]; los grupos R de los aminoácidos se sitúan alternativamente por encima y por debajo de la lámina plegada. Los segmentos adyacentes que forman la lámina β suelen estar próximos en la estructura primaria de la proteína, pero pueden también situarse en regiones alejadas, e incluso pueden pertenecer a diferentes cadenas polipeptídicas.
Además, dos hebras adyacentes pueden seguir la misma dirección (disposición paralela) o direcciones opuestas (antiparalela). La fibroína o β-queratina de la seda es una proteína típica que presenta esta estructura.
- Giros β. Son estructuras secundarias en forma de U, formadas por cuatro restos de aminoácido y esta- bilizadas por un enlace de hidrógeno que se establece entre el –C=O del primer aminoácido y el –NH del cuarto.
Abundan en las regiones de la cadena polipeptídica en las que se da un cambio brusco de dirección; su nombre obedece a que son típicas de las zonas de conexión de los extremos sucesivos de dos segmentos de una proteína con conformación β-laminar (lógicamente, en este caso, los segmentos estarán dispuestos de modo antiparalelo).
Pulsa sobre la imagen para verla ampliada Esquema representativo de una lámina β (fuente: ASH). -
- Estructuras supersecundarias
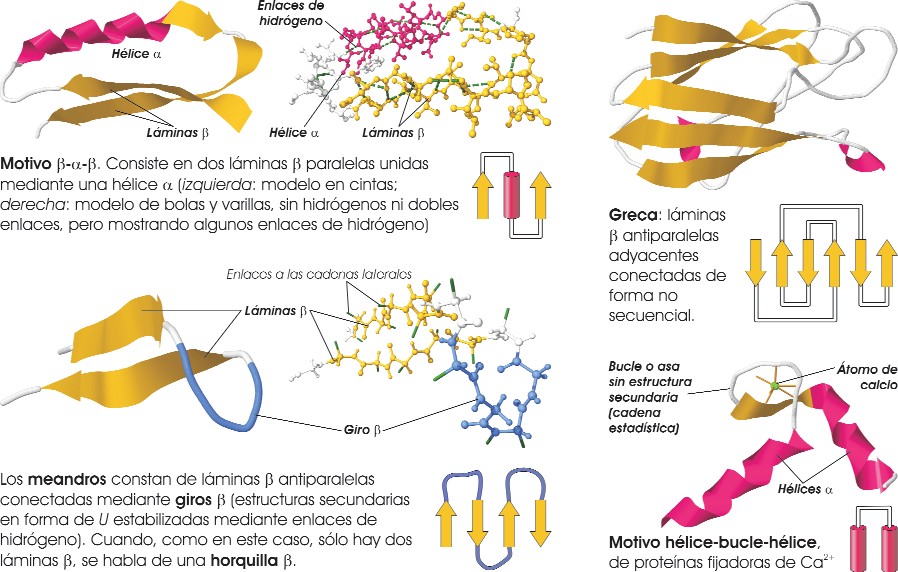
Conocidas frecuentemente como motivos estructurales, se trata de agrupaciones muy estables de varias estructuras secundarias y de las conexiones entre ellas. Se hallan en numerosas proteínas; a menudo un motivo se repite en una misma proteína, y se conocen motivos que engloban a una proteína entera. Son típicos los motivos βαβ, los meandros, las grecas y los motivos hélice-bucle-hélice:
Pulsa sobre la imagen para verla ampliada Estructuras supersecundarias: motivos βαβ, meandros, grecas y motivos hélice-bucle-hélice (fuente: ASH). - Estructura terciaria
Es la disposición tridimensional global de todos los átomos de una cadena polipeptídica. Mientras que la estructura secundaria alude al ordenamiento de restos de aminoácidos adyacentes en la secuencia de aminoácidos, en la estructura terciaria se dan interacciones de largo alcance, esto es, entre aminoácidos muy alejados en la estructura primaria (y alojados quizá en diferentes estructuras secundarias). Dichas interacciones, que estabilizan la estructura terciaria, no involucran a los grupos –C=O y –NH de los enlaces peptídicos, sino a las cadenas laterales R; y tampoco se limitan a enlaces de hidrógeno, sino que incluyen:
- Enlaces covalentes, que pueden deberse a la formación de un enlace disulfuro entre los grupos –SH de dos residuos de cisteína, gracias a la reacción:
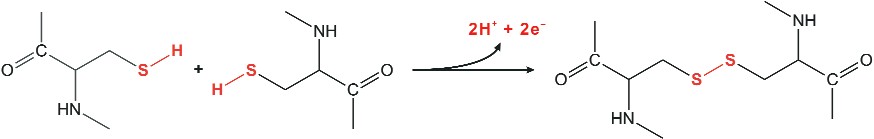
o a la formación de un enlace amida (–CO–NH–) entre el grupo –NH3+ de la cadena lateral de la lisina y el grupo –COO¯ del ácido aspártico o del glutámico.
- Enlaces no covalentes, que pueden ser de tres tipos:
- Fuerzas electrostáticas entre cadenas laterales ionizadas con cargas de signo opuesto.
- Enlaces de hidrógeno entre cadenas laterales polares pero no iónicas.
- Interacciones hidrofóbicas y fuerzas de Van der Waals entre cadenas laterales apolares que estén suficientemente próximas (ya que se trata de interacciones muy débiles).
- Enlaces covalentes, que pueden deberse a la formación de un enlace disulfuro entre los grupos –SH de dos residuos de cisteína, gracias a la reacción:
- Dominios
Las cadenas polipeptídicas de gran tamaño se dividen a menudo en diferentes regiones conocidas como dominios estructurales, que suelen desempeñar funciones distintas (por ejemplo, un dominio puede ser el responsable de la actividad catalítica de una proteína, en tanto que otro se encarga de la fijación de ciertas moléculas). Cada dominio consta de 40 a 350 restos de aminoácidos organizados en varias estructuras secundarias o supersecundarias y se pliega independientemente del resto a medida que se sintetiza la cadena polipeptídica. La disposición de los dominios de una proteína, cada uno con su propia estructura terciaria, se conoce a veces como su estructura superterciaria.
Muchos dominios mantienen su estructura tridimensional correcta incluso cuando se separan del resto de la cadena polipeptídica, lo que ha facilitado el intercambio artificial de dominios entre varias proteínas para construir proteínas quiméricas. Este proceso parece haber sucedido también de forma natural: numerosas proteínas muestran señales de haber evolucionado a través de la unión de dominios preexistentes que se han “barajado” entre sí, generando nuevas combinaciones. Ciertos dominios particularmente móviles a lo largo de la evolución reciben el nombre de módulos proteínicos.
- Estructura cuaternaria
Diversas proteínas constan de una sola cadena polipeptídica, pero otras, conocidas como proteínas con subunidades múltiples, poseen varios polipéptidos asociados por enlaces no covalentes —a veces, como ocurre en las inmunoglobulinas o anticuerpos, los polipéptidos individuales se unen mediante enlaces disulfuro; entonces no se consideran subunidades y se denominan simplemente cadenas—. La disposición de los polipéptidos en complejos tridimensionales constituye la estructura cuaternaria de la proteína.
Según el número de subunidades diremos que la proteína es un dímero (si posee dos), un trímero (si tiene tres), un tetrámero, un pentámero… A menudo la proteína consta de una subunidad que se repite llamada protómero, formada a su vez por una o varias cadenas polipeptídicas. Por ejemplo, la hemoglobina (véase la ilustración siguiente) puede considerarse, bien como un tetrámero de cuatro subunidades polipeptídicas — dos cadenas α y dos β—, bien como un dímero de protómeros αβ.
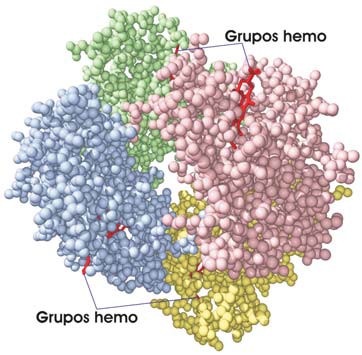
La hemoglobina es una proteína con estructura cuaternaria: incluye dos cadenas denominadas α y otras dos β, unidas cada una a un grupo hemo (fuente: ASH).
Obra publicada con Licencia Creative Commons Reconocimiento No comercial Compartir igual 4.0