3.2. La síntesis de proteínas
Una vez descifrado el código genético, quedaba por aclarar el mecanismo de la traducción. Como sabemos, los codones de una molécula de ARNm no reconocen directamente los aminoácidos que especifican; es necesaria una compleja maquinaria en la que toman parte los ARNt, los ribosomas y una variada colección de enzimas.
Los ribosomas
Los responsables directos de la síntesis de proteínas son unas pequeñas partículas compactas, de forma algo oblonga y con unas dimensiones aproximadas de entre 15 y 25 nm: los ribosomas.
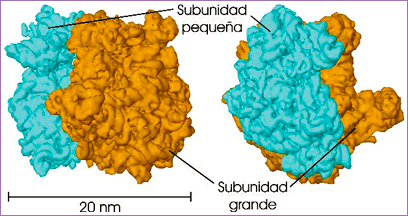 |
|
| Representación esquemática de la superficie molecular de un ribosoma, mostrando las subunidades grande y pequeña (fuente: ASH). |
Los ribosomas carecen de membrana y solo se pueden observar con el microscopio electrónico. Habitualmente se asocian en hileras formadas por diez o más, llamadas polisomas (síncopa de polirribosomas). Existen dos clases de ribosomas: los que se hallan dispersos a millones por el citosol o adheridos al retículo endoplasmático de las células eucarióticas son ribosomas 80S, mientras que en el interior de las bacterias, mitocondrias y cloroplastos se hallan los ribosomas 70S (la “S” representa el svedberg, una unidad que indica la velocidad a la que sedimentan al centrifugarlos, y que se relaciona con su tamaño).
Cada ribosoma se compone de más de cincuenta proteínas y varias moléculas de un ácido nucleico, el ARN, organizadas en una subunidad grande y otra pequeña (ilustración anterior). Los ribosomas reciben instrucciones del ARNm que indican con precisión qué aminoácidos tienen que engarzar y en qué orden; es decir, en estos orgánulos tiene lugar la traducción.
La maquinaria de traducción
El ARNt depende críticamente, para llevar a cabo su función, del plegamiento adecuado, debido a enlaces de hidrógeno. Se forma así una estructura compacta a modo de “L”. En uno de sus lados se halla el anticodón (véase la ilustración siguiente), que puede formar pares de bases con un codón complementario; por el otro lado —en el extremo 3’ de la molécula—, se une al aminoácido correspondiente al codón.
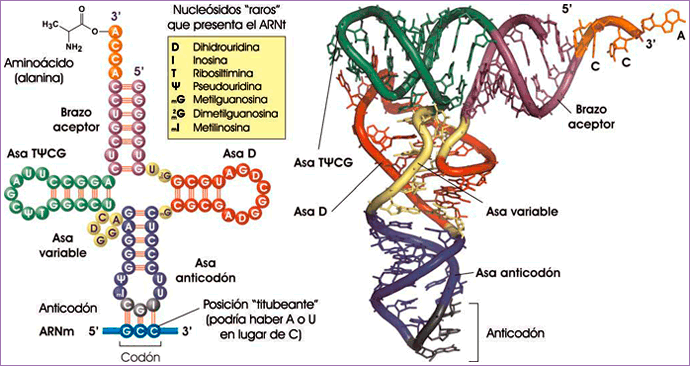
Izquierda: Estructura en “hoja de trébol” de un ARNt, un convenio usado para mostrar zonas donde se aparean bases complementarias de una misma cadena (brazos) y donde no lo hacen (asas); el nombre de dos de ellas remite a la presencia de bases modificadas químicamente cuando el ARNt ya se ha sintetizado. El brazo aceptor es el que se une al aminoácido, y el anticodón es la secuencia de tres nucleótidos que se enlaza con un codón. Derecha: Estructura real (tridimensional) de la molécula, en forma de “L” (fuente: ASH y http://en.wikipedia.org/wiki).
Si la complementariedad entre codones y anticodones fuese perfecta debería haber tantos tipos de ARNt como codones con sentido. Sin embargo, algunos ARNt solo requieren que se emparejen correctamente las dos primeras bases del codón, tolerando cierto titubeo (en inglés, wobble) en la tercera posición. Por ejemplo, el anticodón AAG (escrito en sentido 3’ → 5’) puede reconocer los codones UUC y UUU (ambos en sentido 5’ → 3’). A veces en la posición de titubeo del ARNt hay una guanosina desaminada, la inosina, capaz de aparearse con A, C y U, lo que facilita el reconocimiento de varios codones (ilustración anterior). Esto explica por qué muchos codones sinónimos solo se distinguen en la tercera posición y, por lógica, debería permitir que hubiese menos tipos de ARNt que codones. Curiosamente, ocurre lo contrario: en el ser humano, por ejemplo, hay 497 genes de ARNt —sin incluir los mitocondriales—, aunque entre todos ellos solo hay representados 48 anticodones diferentes.
Aunque el ARNt actúa como el adaptador postulado por Crick, en realidad se necesita otro adaptador, la llamada sintetasa del aminoacil-ARNt, que acopla un aminoácido a su correspondiente ARNt (proceso llamado activación del aminoácido). Suele haber una de tales enzimas para todos los ARNt que se unen al mismo aminoácido:
Aminoácido + ARNt + ATP Sintetasa específica→ Aminoacil-ARNt + AMP + PPi
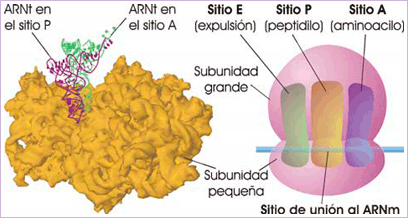 |
|
| Modelo tridimensional (izquierda) y esquema simplificado (derecha) de las subunidades de un ribosoma y sus diferentes sitios de unión a los ARNt (fuente: ASH). |
Los aminoacilARNt así formados se unen a centros específicos de los ribosomas designados como sitios A, P y E (ilustración derecha). En los sitios A y P se puede unir una molécula de ARNt solo si su anticodón está apareado con un codón complementario del ARNm (el cual también se une al ribosoma por un centro específico); ambos sitios se disponen de manera que las dos moléculas de ARNt se vean obligadas a emparejarse con codones adyacentes del ARNm, lo que mantiene el marco de lectura correcto. La determinación, en 2000, de la estructura completa del ribosoma —quizá el mayor logro de la biología molecular— permitió comprobar que los sitios A, P y E están formados básicamente por ARNr, y confirmó la sospecha previa de que son los ARNr, y no las proteínas, los responsables de la actividad ribosómica, incluida su habilidad para formar enlaces peptídicos (actividad enzimática conocida como transferasa del peptidilo). Estas moléculas de ARN con actividad catalítica se llaman ribozimas.
En una misma molécula de ARNm pueden coincidir varios ribosomas, formando polisomas, separados por distancias de 80 nucleótidos. Cada ribosoma se une al extremo 5’ del ARNm, recorre la molécula generando la proteína (lo que lleva entre 20 y 120 segundos) y se desensambla en sus subunidades por el extremo 3’. El proceso se acelera gracias a proteínas que unen los extremos 5’ y 3’ del ARNm, haciendo que los polisomas sean circulares, como se ve en la siguiente ilustración:
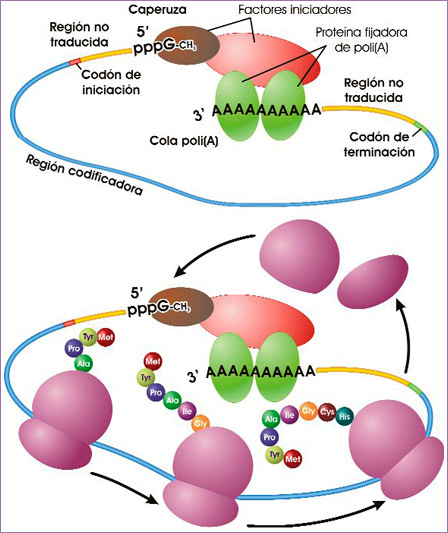
Arriba: Estructura del ARNm de una célula eucariota, con los extremos 5’ y 3’ unidos. Abajo: Polisoma en el que las subunidades ribosómicas, una vez acabada la traducción, hallan fácilmente el extremo 5’ del ARNm y pueden comenzar otra ronda rápidamente (fuente: ASH).
Obra publicada con Licencia Creative Commons Reconocimiento No comercial Compartir igual 4.0
