4.1. Principales técnicas de la ingeniería genética
▼
La ingeniería genética utiliza las denominadas endonucleasas de restricción o restrictasas, unas enzimas que escinden el ADN en puntos concretos para, de esta manera, obtener los segmentos que nos interese. La mayoría de las restrictasas actúan sobre secuencias palindrómicas, es decir, “capicúas”:
5´−G ↓ AATTC → 5´−G AATTC − 3´
3´−C TTAA ↓ G → 3´CTTAA G − 5´
Las flechas indican el punto de corte. Vemos que tras el corte queda un oligonucleótido (en el ejemplo formado por un solo nucleótido) en cada uno de los lados, que son complementarios; estos extremos se llaman cohesivos y facilitan la unión entre distintos fragmentos cortados por la misma restrictasa.
Las enzimas de restricción permiten obtener ADN recombinante in vitro y producir múltiples copias de un determinado gen (proceso denominado clonación). Para ello se ha de aislar el gen e introducirlo en un organismo (por ejemplo, una bacteria) usando un vector adecuado; la célula lo integra en su material genético y, a partir de ese momento, cada vez que se reproduzca se obtiene una nueva copia del gen.
Dichos vectores son moléculas de ADN que se replican independientemente (o bien se integran en el genoma del huésped) y poseen algún rasgo (marcador) que permite que sean fácilmente detectables. También han de presentar al menos un sitio de unión a la restrictasa. Los principales vectores utilizados en ingeniería genética son:
- Plásmidos. Son fragmentos de ADN relativamente pequeños que suelen llevar genes de resistencia específicos a uno o más antibióticos y varios sitios de unión para las restrictasas.
- Virus. Análogamente a lo que ocurre en la naturaleza, el ADN foráneo se introduce dentro de la cápsida. Suelen tener varios lugares de unión para las endonucleasas.
- Cósmidos, de origen artificial. Se asemejan a plásmidos y se comportan como tales en las células, pero son más grandes (lo que permite transportar muchos genes).
- Los YAC (del inglés yeast artificial chromosomes, “cromosomas artificiales de levaduras”), que contienen todos los elementos necesarios para la replicación autónoma de un cromosoma (origen de replicación, centrómeros y telómeros). Con los YAC se pueden insertar y clonar moléculas de ADN de cientos de miles de bases. Se usan cuando se han de clonar genomas muy grandes, como en el caso humano.
Los principales pasos de la clonación del ADN son:
- Aislamiento del ADN que queremos clonar (ADN pasajero) y selección del vector mediante enzimas de restricción. Ambos deben presentar extremos cohesivos.
- Unión del fragmento de ADN al vector por la ligasa del ADN. Esta enzima establece los enlaces fosfodiéster y se obtienen moléculas híbridas o recombinantes:
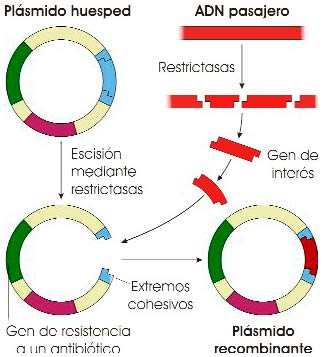
Obtención de un plásmido recombinante (fuente: ASH). - Introducción del recombinante en la célula hospedadora [véase la ilustración 7.24]. Este proceso es muy variable, en función de la célula hospedadora. Puede ocurrir por transformación, transferencia, conjugación (gracias a los pili), fusión de protoplastos que posteriormente se dividen, microinyección…
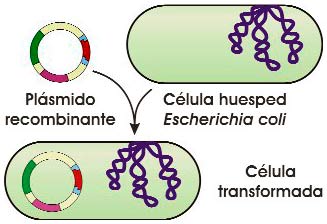
Introducción del plásmido recombinante en la célula huésped (fuente: ASH). - Localización de las células que han captado las moléculas recombinadas. Es el paso más laborioso y donde juegan un papel importante los marcadores presentes en el vector; estos marcadores pueden ser determinados genes (por ejemplo, los que confieren resistencia a un determinado antibiótico, como se muestra la ilustración 7.25) o sondas marcadas radiactivamente (una sonda es una molécula de ADN de cadena sencilla y complementaria del ADN que nos interesa localizar).
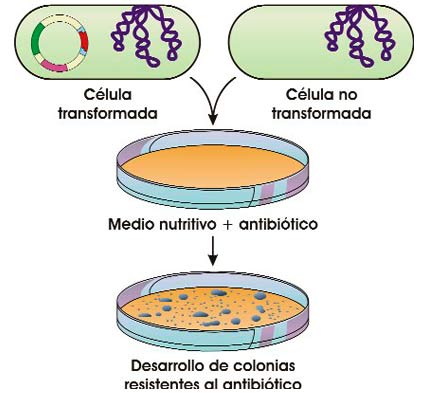
Selección de colonias recombinantes: en un medio nutritivo al que se le ha añadido un determinado antibiótico se hacen crecer bacterias no transformadas y bacterias transformadas (tienen el gen que confiere resistencia al antibiótico). Solo estas últimas crecen y forman colonias; las no transformadas mueren (fuente: ASH). - Por último, se ha de determinar si la información genética aportada por las moléculas recombinantes se mantiene estable en las siguientes generaciones.
El objetivo final es la obtención de al menos una colonia (clon) de células que lleven el recombinante, en cuyo caso, se ha logrado clonar, aislar, el ADN pasajero.
Las bacterias eran los organismos que comúnmente se usaban en los comienzos de la ingeniería genética. Hoy en día la manipulación se extiende a todo tipo de organismos, incluidos los animales y las plantas.
▼
▼
 |
|
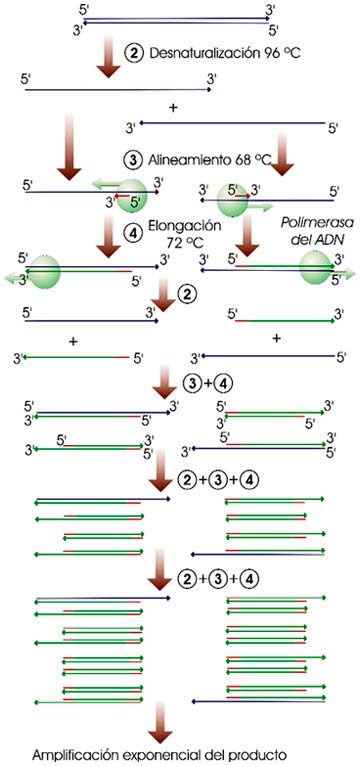
| Esquema de sucesivos ciclos de la PCR. Estos ciclos se repiten hasta obtener el número de copias deseado (fuente: www.wikipedia.org/wiki). |
Las técnicas del ADN recombinante tienen varios inconvenientes: su lentitud, la necesidad de efectuarse en un ser vivo y la escasa cantidad de ADN recombinante que se obtiene. La conocida como reacción en cadena de la polimerasa de ADN (PCR, de polymerase chain reaction) ha venido a solventar estas dificultades, al permitir generar, en pocas horas, millones de moléculas idénticas a partir de una sola molécula de ADN. Esta técnica exige la síntesis previa de unos oligonucleótidos específicos que hibriden con el principio y el final del fragmento que se desea amplificar, actuando como ADN cebador (primer). La PCR consta de varios pasos (ilustración adjunta):
- Se incuba en un tubo de ensayo la muestra de ADN con la enzima polimerasa, trifosfatos de nucleósido y los dos oligonucleótidos cebadores.
- Se calienta el tubo a 96 ºC durante 5 minutos; las dos hebras de ADN se separan.
- Se reduce la temperatura a unos 68 ºC durante 1 minuto; los cebadores se unen.
- La temperatura se incrementa a 72 ºC durante 5 minutos, para que actúe la polimerasa y se formen las cadenas complementarias.
- Se sube la temperatura a 94 ºC durante 20 segundos, suficientes para separar la cadena recién sintetizada del molde original.
Las cadenas sencillas generadas entran en sucesivos nuevos ciclos (repetición de los pasos 1 al 5); tras 30 a 60 ciclos el ADN original se ha amplificado millones de veces.
La PCR se ha convertido en una técnica indispensable en gran variedad de aplicaciones: en clínica (como método de diagnóstico), en medicina forense (para determinar si una muestra orgánica pertenece o no a un individuo), en paleontología molecular (secuencias de organismos extinguidos cuyo ADN se ha conservado se pueden amplificar, secuenciar y comparar con especies contemporáneas)…, porque permite obtener una gran cantidad de ADN para analizar.
▼
▼
Uno de los aspectos más impactantes y populares de la ingeniería genética es la identificación de la huella genética o perfilado del ADN, utilizado en exámenes forenses para la identificación de personas y pruebas de paternidad a partir de una pequeña muestra orgánica (sangre, semen, pelos…).
La base científica de la huella genética se halla en la existencia de regiones no codificantes del ADN llamadas minisatélites o VNTR (siglas de variable number of tandem repeats, “número variable de repeticiones en tándem”) repartidas por todo el genoma humano. Una repetición en tándem es una secuencia corta de ADN que se repite consecutivamente en un locus cromosómico específico. El número exacto de repeticiones varía entre individuos y, por lo tanto, varía la longitud de las VNTR.
Para obtener estas regiones se utilizan técnicas del ADN recombinante. También se puede utilizar la PCR si el tamaño de la muestra es muy pequeña; los fragmentos obtenidos se separan por electroforesis (véase la ilustración siguiente). La utilización de sondas radiactivas permite la localización de los VNTR.

El análisis de un locus VNTR mediante hibridación suele mostrar un patrón de dos bandas, uno heredado del padre y otra de la madre. Puede darse un patrón de una sola banda, si el tamaño de las dos bandas es el mismo o muy similar. Habitualmente, los perfiles de un solo locus VNTR de individuos no relacionados entre sí son diferentes; no obstante, es posible que dos personas tengan el mismo perfil en uno o dos loci, aunque las posibilidades de coincidencia disminuyen drásticamente a medida que se incrementa el número de loci comparados. Cuando se usan los perfiles de ADN con fines medicolegales, se analizan generalmente de 4 a 6 loci VNTR diferentes.
▼
Obra publicada con Licencia Creative Commons Reconocimiento No comercial Compartir igual 4.0
