3.1. El código genético
Se ha comentado ya que la interpretación de los “planos” de ARNm por parte de los ribosomas es básicamente un proceso de traducción. Y como todo traductor, el ribosoma debe disponer de un diccionario. Recordemos (véase la actividad 7) que las entradas del mismo deben ser combinaciones de tres nucleótidos: las combinaciones binarias (con repetición) generarían 16 palabras, o sea, solo permitirían la incorporación de 16 aminoácidos a las proteínas. Crick propuso el término codón para designar a cada uno de los 64 tripletes de nucleótidos del ARNm y formuló una pregunta fundamental: ¿cómo averiguar en qué términos está redactado el diccionario?
El código “sin comas”
El propio Crick sugirió una respuesta que, en realidad, era una alternativa al fallido código de Gamow. Crick se dio cuenta de que una secuencia de ARNm podría traducirse siguiendo tres posibles marcos de lectura distintos, según se explica en la ilustración siguiente. ¿Cómo reconocerían las células cuál es la pauta correcta? ¿Acaso existirían “signos de puntuación” especiales (comas) que delimitasen los codones verdaderos evitando lecturas alternativas?
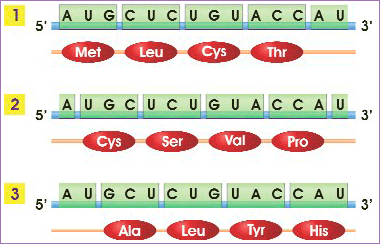 Un código sin comas podría dar lugar a distintos marcos de lectura: el mismo ARNm especificaría tres secuencias de aminoácidos, según dónde comenzase la lectura (fuente: ASH).
Un código sin comas podría dar lugar a distintos marcos de lectura: el mismo ARNm especificaría tres secuencias de aminoácidos, según dónde comenzase la lectura (fuente: ASH).El modelo de Crick eludía la necesidad de comas con un diccionario de palabras sin sentido, que limitaba el número de codones que cifran aminoácidos. Si, en el ejemplo de la ilustración anterior, AUG3 y CUC codificasen aminoácidos (Met y Leu, respectivamente), se evitaría que un cambio del marco de lectura originase un polipéptido incorrecto si UGC y GCU careciesen de sentido, esto es, si no definiesen ningún aminoácido —en la ilustración, en cambio, sí lo hacen—. Un cálculo sencillo mostraba que solo podrían existir, en tal caso, 20 codones con sentido… justo los correspondientes a los 20 aminoácidos identificados por entonces. Los 44 codones restantes serían codones sin sentido.
El descifrado del código genético
El modelo de Crick era tan estilizado y coherente que se granjeó la aceptación casi universal… hasta el punto de ser calificado como “la más elegante teoría biológica que ha resultado ser incorrecta”. Efectivamente, la naturaleza parece haber optado por un esquema menos “perfecto”. Su escudriñamiento se remonta a 1955, cuando la bioquímica franco-rusa Marianne Grunberg-Manago (n. 1921) aisló a partir de extractos bacterianos una enzima llamada polinucleótido fosforilasa (PNPasa). La PNPasa cataliza la hidrólisis del ARN, pero también puede sintetizar ARN a partir de una mezcla de difosfatos de ribonucleósido que engarza al azar, sin necesidad de molde previo, lo cual posibilita la obtención in vitro de ARNm cuyas secuencias estén en cierto modo controladas.
Con auxilio de la PNPasa el bioquímico estadounidense Marshall Warren Nirenberg (n. 1927) y el alemán Johann Heinrich Matthaei (n. 1929) sintetizaron en 1961 un ARN formado únicamente por nucleótidos de uracilo —un poli(U)— y lo añadieron a un sistema libre de células: un tubo de ensayo que solo contenía ribosomas, enzimas, ARNt, aminoácidos y ATP como fuente de energía. Se originó un polipéptido formado por una sucesión de restos del aminoácido fenilalanina (Phe); puesto que el único codón posible en el poli(U) era UUU, estaba claro que, en el diccionario genético, UUU = Phe. Ensayos análogos demostraron que el triplete CCC codifica para prolina (Pro) y AAA para la lisina (Lys); es decir, hasta los codones que según Crick presuntamente carecían de significado [véase la actividad 11] podían provocar la síntesis de proteínas. Más adelante, el biólogo de origen hindú Har Gobind Khorana (n. 1922) empleó ARNm sintéticos compuestos por nucleótidos alternantes (véanse las actividades 13 y 14). Con este y otros tipos de métodos, en unos cuantos años se pudo desvelar la totalidad del código.
Obra publicada con Licencia Creative Commons Reconocimiento No comercial Compartir igual 4.0
