1.2.1. El replisoma de las bacterias
La ADN pol III recuerda a la polimerasa del ARN propia de la transcripción: ambas polimerizan en sentido 5’ → 3’ y requieren un molde de ADN; la formación de enlaces fosfodiéster está favorecida en los dos casos por la posterior hidrólisis del pirofosfato, debida a la enzima piro-fosfatasa. No obstante, existen también diferencias notables entre ambas:
- La polimerasa del ADN une desoxirribonucleótidos, no ribonucleótidos.
- La polimerasa del ADN “lee” las dos hebras del ADN, no una sola.
- La polimerasa del ADN presenta actividad exonucleasa en sentido 3’ → 5’, es decir, elimina el último nucleótido incorporado si no es complementario. Esta capacidad para corregir errores falta en la polimerasa del ARN, razón por la cual la tasa de error en la transcripción es mayor que en la replicación.
- La polimerasa del ADN es incapaz de desenrollar el ADN y separar las dos cadenas que deben copiarse. La polimerasa del ARN, en cambio, sí lo hace.
- Ni la ADN pol III ni ninguna otra polimerasa del ADN es capaz de iniciar la síntesis; tan solo puede alargar una cadena (un polinucleótido) ya existente.
La complejidad enzimática del replisoma (véase la ilustración a continuación) refleja precisamente la necesidad de solucionar los problemas de desenrollamiento, iniciación y síntesis discontinua en la hebra retrasada.
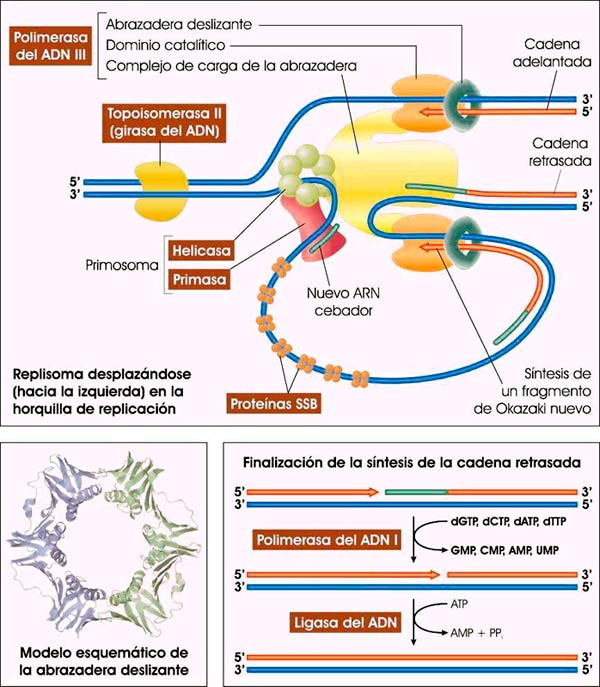
Principales tipos de proteínas que actúan en la horquilla de replicación de las bacterias (falta añadir la pirofosfatasa, citada en la ilustración 7.6). Obsérvese que el ADN se pliega sobre la cadena retrasada para permitir que una sola molécula de ADN pol III, con dos subunidades catalíticas, sintetice a la vez las hebras adelantada y retrasada (fuente: ASH).
El proceso incluye varios acontecimientos:
- A la región de origen del cromosoma, identificada como oriC, se unen unas proteínas iniciadoras que separan las hebras de ADN gracias a la energía de hidrólisis de ATP. El complejo de proteína y ADN se une a dos anillos llamados helicasas, que desenrollan el ADN en ambos sentidos y crean dos horquillas de replicación.
- Numerosas proteínas SSB (del inglés single-stranded DNA binding, “unión a ADN de una sola cadena”) se asocian a las cadenas de ADN separadas por la helicasa e impiden que se vuelvan a unir, permitiendo así la actuación de la polimerasa.
- La separación de las hebras en la horquilla de replicación crea tensiones tras esta, que pueden originar superenrollamientos; la girasa del ADN es la encargada de relajarlas.
- Para que la polimerasa del ADN pueda actuar es necesario un cebador, esto es, una corta cadena a cuyo extremo 3’ pueda añadir nucleótidos. El cebador (primer, en inglés) es un oligonucleótido de ARN, no de ADN, y se sintetiza por una enzima llamada primasa, que se asocia a la helicasa formando un primosoma.
- En cada horquilla, la ADN pol III cataliza la síntesis continua de la cadena adelantada y, simultáneamente, de un fragmento de Okazaki a partir de un cebador. Para evitar que la ADN pol III se disocie del ADN, incluye subunidades en forma de rosquilla llamadas abrazaderas deslizantes.
- Una vez formado cada fragmento de Okazaki, la ADN pol I elimina los ribonucleótidos del cebador gracias a que posee actividad exonucleasa 5’ → 3’ (además de la actividad exonucleasa 3’ → 5’ típica de las polimerasas de ADN) y los reemplaza por los correspondientes desoxirribonucleótidos gracias a su actividad polimerizadora.
- Tras la eliminación del cebador y el relleno del hueco con ADN aún queda una mella entre el extremo 3’ de cada nuevo fragmento de ADN y el extremo 5’ del anterior. La ligasa del ADN sella entonces dicho hueco formando un enlace fosfodiéster.
- Cuando las dos horquillas de replicación iniciadas en oriC se aproximan al extremo opuesto del cromosoma circular de Escherichia coli tropiezan con la secuencia Ter (de terminador) que detiene su avance. Finalmente quedan dos cromosomas circulares encadenados, que más tarde se separan.
La replicación del cromosoma bacteriano está asociada a la membrana plasmática: la secuencia oriC es “secuestrada” por un complejo formado por dos replisomas (véase la ilustración a continuación) anclados a la membrana en el centro de la célula; tras duplicarse, los dos puntos oriC se desplazan a extremos opuestos y “arrastran” así cada cromosoma a su correspondiente célula hija.
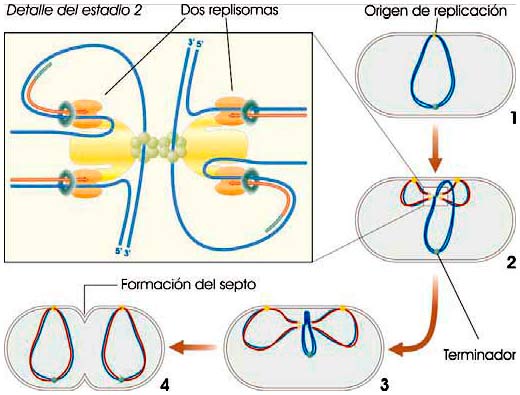
En la replicación del cromosoma bacteriano no es el replisoma el que avanza a lo largo de cada horquilla, sino que ambos replisomas permanecen juntos y anclados a la membrana y es el ADN el que avanza a su través (fuente: ASH).
Obra publicada con Licencia Creative Commons Reconocimiento No comercial Compartir igual 4.0
